NVIDIA的VISTA-2D模型革新了细胞成像和空间组学
realtime news Jul 25, 2024 02:47
NVIDIA推出的VISTA-2D模型提升了细胞分割和形态聚类在空间组学中的表现,为生物研究提供了重要的进步。

NVIDIA已经发布了VISTA-2D,一个旨在显著改善细胞成像和空间组学工作流程中细胞分割的基础模型,详见NVIDIA技术博客。该模型利用先进的图像嵌入技术,旨在提高下游任务的准确性。
特征提取和聚类
VISTA-2D模型使用图像编码器生成可以转换为分割掩码的嵌入。这些嵌入提供了关于细胞形态的必要信息,从而实现精确的细胞分割。NVIDIA的博客文章解释了这些嵌入可以通过聚类来自动分组具有相似形态的细胞。
为了展示模型的能力,NVIDIA提供了一个详细的Jupyter notebook,指导用户如何使用VISTA-2D进行细胞分割和提取空间特征。Notebook还展示了如何使用RAPIDS聚类这些特征,从而创建自动化的细胞类型分类管道。
前提条件和设置
对VISTA-2D模型感兴趣的用户需要具备Python、Jupyter和Docker的基本知识。可以通过以下命令启动本教程所需的Docker容器:
docker run --rm -it \
-v /path/to/this/repo/:/workspace \
-p 8888:8888 \
--gpus all \
nvcr.io/nvidia/pytorch:24.03-py3 \
/bin/bash
可以使用以下命令安装本教程所需的其他Python包:
pip install -r requirements.txt
使用VISTA-2D进行细胞分割
初始步骤包括加载VISTA-2D模型检查点并使用它来分割图像中的细胞。分割过程生成每个细胞的特征向量,其中包含进行细胞形态分析所需的所有信息。这些向量然后被用于聚类算法以分组具有相似特征的细胞。
细胞分割
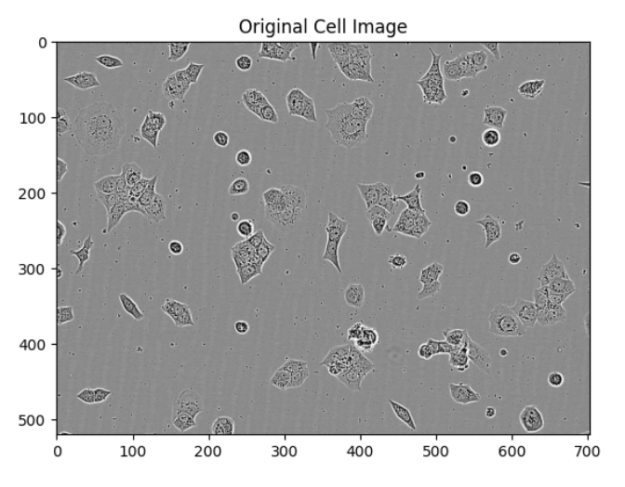
分割函数通过VISTA-2D处理细胞图像,生成标记每个细胞的分割掩码。这允许对每个细胞进行准确的特征提取。
img_path="example_livecell_image.tif" patch, segmentation, pred_mask = segment_cells(img_path, model_ckpt)
绘制分割
可以使用plot_segmentation函数对分割后的图像进行可视化验证。此函数显示原始图像、分割结果和每个细胞的单独掩码。
plot_segmentation(patch, segmentation, pred_mask)


使用RAPIDS进行特征聚类
一旦提取了特征向量,它们将使用RAPIDS进行聚类,这是一种GPU加速的机器学习库。TruncatedSVD算法降低了特征向量的维度,使得更易于在3D空间中可视化聚类结果。
dim_red_model = TruncatedSVD(n_components=3) X = dim_red_model.fit_transform(cell_features)
然后使用DBSCAN算法对降维后的特征向量进行聚类。这种方法为每个细胞分配聚类标签,可以使用Plotly进行互动3D图可视化。
model = DBSCAN(eps=0.003, min_samples=2) labels = model.fit_predict(X)

结论
NVIDIA的VISTA-2D模型通过提供精确的细胞分割和特征提取,为细胞成像和空间组学提供了显著进步。结合RAPIDS进行聚类,该模型实现了细胞类型的高效分类,为更详细和自动化的生物研究铺平了道路。
Image source: Shutterstock.jpg)